Permanent·e·s: Emmanuelle Bignon (CR CNRS), Christophe Chipot (DR CNRS & Adjunct professor of physics University of Illinois at Urbana-Champaign), François Dehez (DR CNRS), Sébastien Fumeron (MCF HDR), Thérèse Malliavin (DR CNRS), Martin Müller (MCF HDR), Mounir Tarek (DR CNRS)
Non-permanent·e·s: Anna Ananchenko (doctorante), Sergio Arredondo-Contreras (postdoc), Giuseppe Chen (postdoc), Roy Gonzales-Aleman (postdoc), Mayank Pandey (postdoc), Florence Szczepaniak (doctorante), Qinfan Wang (postdoc)
Les approches théoriques sont aujourd’hui incontournables pour décrire précisément la biophysique et la biochimie de processus biologiques fondamentaux. L’axe de recherche Biochimie et Biophysique fédère l’ensemble des travaux de physique et de chimie du laboratoire LPCT centrés sur la modélisation du « vivant ». Ceux-ci portent sur l’étude de cibles communes (membranes biologiques, protéines, acides nucléiques) à différentes échelles de taille et de temps (réactivité enzymatique, transport membranaire, croissance de plantes, …) et sur le développement d’approches théoriques innovantes permettant de repousser les limites actuelles.
Publications récentes:
Szczepaniak, F., Dehez, F., & Roux, B. (2024). Configurational Sampling of All-Atom Solvated Membranes Using Hybrid Nonequilibrium Molecular Dynamics Monte Carlo Simulations. The Journal of Physical Chemistry Letters, 15(14), 3796-3804.
Delort, A., Cottone, G., Malliavin, T. E., & Müller, M. M. (2024). Conformational Space of the Translocation Domain of Botulinum Toxin: Atomistic Modeling and Mesoscopic Description of the Coiled-Coil Helix Bundle. International Journal of Molecular Sciences, 25(5), 2481.
Karami, Y., & Bignon, E. (2024). Cysteine hyperoxidation rewires communication pathways in the nucleosome and destabilizes the dyad. Computational and Structural Biotechnology Journal, 23, 1387-1396.
Rems, L., Rainot, A., Wiczew, D., Szulc, N., & Tarek, M. (2024). Cellular excitability and ns-pulsed electric fields: Potential involvement of lipid oxidation in the action potential activation. Bioelectrochemistry, 155, 108588.
Szlasa, W., Sauer, N., Baczyńska, D., Ziętek, M., Haczkiewicz-Leśniak, K., Karpiński, P., Fleszar, M., Fortuna, P., Kulus, M. J., Piotrowska, A., Kmiecik, A., Barańska, A., Michel, O., Novickij, V., Tarek, M., Kasperkiewicz, P., Dzięgiel, P., Podhorska-Okołów, M., Saczko, J., & Kulbacka, J. (2024). Pulsed electric field induces exocytosis and overexpression of MAGE antigens in melanoma. Scientific Reports, 14(1), 12546.
Cournia, Z., & Chipot, C. (2024). Applications of Free-Energy Calculations to Biomolecular Processes. A Collection. Journal of Chemical Information and Modeling, 64(7), 2129-2131.
Goulard Coderc de Lacam, E., Roux, B., & Chipot, C. (2024). Classifying Protein–Protein Binding Affinity with Free-Energy Calculations and Machine Learning Approaches. Journal of Chemical Information and Modeling.
López-Sánchez, U., Munro, L. J., Ladefoged, L. K., Pedersen, A. J., Brun, C. C., Lyngby, S. M., Baud, D., Juillan-Binard, C., Grønborg Pedersen, M., Lummis, S. C. R., Bang-Andersen, B., Schiøtt, B., Chipot, C., Schoehn, G., Neyton, J., Dehez, F., Nury, H., & Kristensen, A. S. (2024). Structural determinants for activity of the antidepressant vortioxetine at human and rodent 5-HT3 receptors. Nature Structural & Molecular Biology, 1-11.
Gillet, N., Dumont, E., & Bignon, E. (2024). DNA damage and repair in the nucleosome: insights from computational methods. Biophysical Reviews, 1-12.
Smirnova, E., Bignon, E., Schultz, P., Papai, G., & Shem, A. B. (2024). Binding to nucleosome poises human SIRT6 for histone H3 deacetylation. Elife, 12, RP87989.
Thèmes scientifiques :
Nouvelles méthodes statistiques et quantiques pour la modélisation de systèmes biologiques complexes
Depuis presque quarante ans, la dynamique moléculaire, sorte de microscope informatique, est largement utilisée dans l’étude d’objets biologiques, formant ainsi un complément précieux des approches classiques de biologie structurale. De nombreux processus cellulaires restent cependant hors d’atteinte pour cette approche numérique, compte tenu des échelles de temps qu’ils couvrent. Au regard de la complexité des objets en jeu, différentes alternatives existent pour échantillonner les degrés de liberté caractéristiques les plus lents.
Nous développons des méthodes à la croisée des schémas d’échantillonnage d’importance et d’ensembles généralisés susceptibles d’offrir une solution convaincante aux limitations de la dynamique moléculaire. Dans le cadre du Laboratoire International Associé CNRS-UIUC , qui lie le CNRS à l’Université d’Illinois, ces développements sont portés dans les codes populaires NAMD et VMD, et sont utilisés dans l’étude de la thermodynamique de processus biologiques allant du changement conformationnel impliqué dans le transport membranaire aux phénomènes de reconnaissance et d’association de protéines

Croissance, communication cellulaire et réparation génomique

Nous nous intéressons au fonctionnement de processus biologiques fondamentaux à différentes échelles (du moléculaire au tissu) par des approches macro-, méso- et microscopiques. Nous étudions, en particulier, des problèmes concernant la mécanique des membranes biologiques ainsi que la croissance des tissus mous, comme les plantes ou encore les tissus cancéreux essentiellement à l’aide de la mécanique des interfaces fluides et élastiques. Jusqu’à présent les systèmes étudiés étaient en équilibre.
Nous modélisons également les filaments du cytosquelette des cellules et leurs interactions avec les membranes biologiques. Le comportement de ces filaments ne peut pas s’expliquer par la physique des polymères standard. Nous avons montré que des fluctuations conformationnelles internes doivent être prises en compte pour comprendre les microtubules. Cela a donné lieu à la notion de confotronique des biofilaments.
L’investigation par modélisation moléculaire de phénomènes qui contrôlent et modulent des processus clés des membranes biologiques impliqués dans la communication cellulaire est divisée en plusieurs thèmes. Ces travaux concernent le Laboratoire International Associé Pulsed Electric Fields Applications in Biology and Medicine. L’étude des phénomènes d’oxydation des membranes. L’oxydation des lipides des membranes biologiques est un processus clé qui influe sur les organismes vivants et le stress oxydatif des membranes.
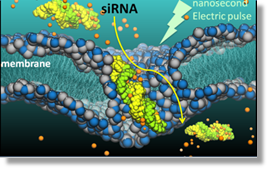

Elle est reliée à l’apparition de diverses maladies allant de l’inflammation de cancer et le vieillissement. L’ultime but de cette caractérisation est de nous permettre de proposer des nouvelles molécules anti-oxydantes membranaires efficaces. La vectorisation par ultrasons. Il s’agit d’utiliser des agents de contraste (microbulles) chargées ou non et des ultrasons pour perméabiliser les cellules avoisinantes. La caractérisation à l’échelle moléculaire de ce phénomène permettra le développement d’une technologie efficace pour la livraison ciblée de molécules.
Les canaux ioniques. L’objectif consiste à mettre en lumière les cascades physiopathologiques conduisant à des arythmies cardiaques et développer de nouveaux peptides pour moduler les canaux ioniques impliqués dans de telles cascades. Les transporteurs membranaires. A l’instar des canaux ioniques, ces protéines permettent d’importer ou d’exporter des espèces chimiques au travers de la membrane. Nous souhaitons ici décrypter la thermodynamique des changements conformationnels associés au fonctionnement de ces protéines pour mieux comprendre leurs dysfonctionnements et les pathologies qui leurs sont associées.


L’ADN est une cible biologique fondamentale au centre notamment des nouvelles techniques d’édition génomique ou encore des traitements par radiothérapie. Pour maintenir l’intégrité de l’information génomique, les cellules ont mis en place des stratégies pour réparer les lésions de l’ADN.
Pour mieux comprendre les mécanismes liés à l’endommagement et à la réparation, nous modélisons l’interaction de brins d’ADN, lésés ou non, avec des protéines de reconnaissance et de réparation. En particulier nous nous intéressons à la compréhension des facteurs structuraux et biophysiques (déformation de l’ADN, stabilité des complexes avec protéines) ainsi qu’à la réactivité sous-jacente, étudiée à l’aide de méthode QM et QM/MM, et à l’étude de la régulation épigénétique.
Collaborations principales :
Nationales
Université de Lorraine (CRM2, CRAN, L2CM, LORIA, IMoPA), ICS (Strasbourg), IBS, SyMMES (Grenoble), ENS (Lyon), IGR (Villejuif)
Internationales
MIT (Boston)
MRC MBU (Cambridge)
Cornell University (New York)
UIUC (Urbana-Champaign)
University of Chicago
Nankai University (Tianjin)
University of Bremen
Hong-Kong Chinese University
University of Pennsylvania
University of Washington
KAIST (South Korea)
Alumni :
Postdoctoral Researchers
• Dr. Wagner Rocha
A local approach for calculating protein conformations: influence of the sampling scheme and of the stereochemistry
2023 (T. Malliavin)
ANR PRCI multiBioStruct
• Dr. Haochuan Chen
Enhanced-sampling methods for free-energy calculations
2022-2023 (C. Chipot)
ANR ProteaseInAction
• Dr. Joel Montalvo Acosta
Structure and Dynamics of the ADP/ATP mitochondrial carrier
2022-2023 (C. Chipot/F. Dehez)
ANR ProteaseInAction
· Dr. Alberto Megias Fernández
A holistic approach to bridge the gap between microsecond computer simulations and millisecond biological events
2023-2024, Financement postdoctoral LUE (C. Chipot)
Graduate Students
• Marharyta Blazhynska
Modeling and standard binding free energy of complex biological objects
2020-2023 (C. Chipot/F. Dehez), defense date 16/10/2023
LUE PhD Research Fellow/ANR ProteaseInAction
• Emma Goulard Coderc de LaCam
Molecular basis of protein protein interactions during synaptogenesis
2020-2023 (C. Chipot), defense date 20/11/2023
LUE PhD Research Fellow/ANR Lorraine Artificial Intelligence
• Tom Miclot
Modeling the influence of DNA lesion on the regulation of gene expression
2019-2022 (A. Monari, G. Barone), defense date 12/12/2022
Codirection Université de Lorraine and Università degli studi (Palerme, Italia)
• Cécilia Hognon
Molecular modeling and simulation to understand the fundamental biological processes and develop novel therapeutic agents against cancer and Covid-19
2018-2021 (A. Monari, S. Grandemange), defense date 11/10/2021
Undergraduate Students
• Yani Kedjar
Étude des mécanismes épigénétiques oxydatifs par modélisation moléculaire
2024, M1 Biologie cellulaire & Physiologie (E. Bignon)
• Larissa Aboudem-Joumessi & Jules Poncelet
Guider la conception d’intercalateurs d’ADN photo-déclenchés grâce à la modélisation moléculaire
2024, M1 Physique (E. Bignon)
• Lynn Buccholz
In silico modeling of lipid membranes
2023, L3 Licence Sciences du Vivant Biochimie-Biologie Moléculaire (F. Szczepaniak/F. Dehez)
Orion fellowship
• Jordan Klein
Modèles physiques décrivant l’interaction des neurotoxines botuliques avec la
membrane de la vésicule synaptique
2023, M2 Physique (T. Malliavin/M. Müller)
• Mailys Duch
Modélisation de composés intercalants de l’ADN : Propriétés structurales et spectrales
2023, L3 Chimie-PCE (E. Bignon)
• Stéphane Ferreira da Costa & Victorien Simon
Simulation de spectres de dichroïsme circulaire de systèmes complexes
2023, M1 Physique (E. Bignon/T. Etienne)
• Zineb Elftmaoui
Élucider le rôle de la S-glutathionylation sur la dynamique de la chromatine à l’aide de la modélisation moléculaire
2023, M1-Sciences du vivant – ORI Biochimie, Biologie Moléculaire et Régulations Cellulaires (E. Bignon)
• Mathieu Merabti
Analyse des relations entre angles de liaison, angles de torsion et longueurs des tri-peptides dans les structures de protéines
2022, L3 Chimie (T. Malliavin)
• Enguerrand Galmiche
Étude de l’interaction entre ADN et Histone (HU) chez Déinococcus radiodurans
2022, M1 Ingénierie de la Santé – Parcours-Type Innovations en Thérapeutique et Diagnostic (F. Dehez/E. Bignon)
• Maxime Petitjean
Utilisation de méthodes multi-échelles pour caractériser l’interaction entre une protéine et de l’ADN.
2022, M1 Physique (E. Bignon/T. Etienne)
• Jérémy Morere
Influence des mutation de STimulator of INterferon Genes, STING, sur sa structure.
2021, M1 Chimie (A. Monari/E. Bignon)
• Charlotte Etienne
Effect of cholesterol on the functional dynamic of the 5-HT3 pentameric ligand-gated ion channel.
2019, M2 Master Sciences du Vivant – Biotechnologies – Génie Cellulaire (F. Dehez)
• Julien Colin
Coarse-grained MD simulations of DNA
2019, M1 Master Sciences Physiques (F. Dehez)
Others
• Aissata Wane
Research Assistant, SEQNEVIR project, 2023 (A. Monari/E. Bignon)
• Monha Pepe
Highschool interships in 2022 and 2023 (E. Bignon)
