Participants: Emmanuelle Bignon (CR CNRS), Christophe Chipot (DR CNRS & Adjunct professor of physics University of Illinois at Urbana-Champaign), F. Dehez (DR CNRS), S. Fumeron (MCF HDR) T. Malliavin (DR CNRS), M. Müller (MCF HDR), M. Tarek (DR CNRS)
Le décryptage au niveau moléculaire du fonctionnement d’assemblages complexes reste une gageure au niveau expérimental. Les approches théoriques sont aujourd’hui incontournables pour décrire précisément la biophysique et la biochimie de processus biologiques fondamentaux. L’axe de recherche Biochimie et Biophysique fédère l’ensemble des travaux de physique et de chimie du laboratoire LPCT centrés sur la modélisation du « vivant ». Ceux-ci portent sur l’étude de cibles communes (membranes biologiques, protéines, acides nucléiques) à différentes échelles de taille et de temps (réactivité enzymatique, transport membranaire, croissance de plantes, …) et sur le développement d’approches théoriques innovantes permettant de repousser les limites actuelles.
Publications récentes:
Perturbations of Native Membrane Protein Structure in Alkyl Phosphocholine Detergents: A Critical Assessment of NMR and Biophysical Studies
C. Chipot; F. Dehez; J. R. Schnell; N. Zitzmann; E. Pebay-Peyroula; L. J. Catoire; B. Miroux; E. R. S. Kunji; G. Veglia; T. A. Cross; P. Schanda;
Chemical Reviews (2018) 118 (7): 3559-3607
α-Helix Unwinding as Force Buffer in Spectrins
H. Takahashi; F. Rico; C. Chipot; S. Scheuring
ACS Nano (2018) 12 (3): 2719-2727
Conformational polymorphism or structural invariance in DNA photoinduced lesions: implications for repair rates
F. Dehez; H. Gattuso; E. Bignon; C. Morell; E. Dumont; A. Monari
Nucleic Acids Research (2017) 24 (7): 1837-3662
Crystal Structure of the DFNKF Segment of Human Calcitonin Unveils Aromatic Interactions between Phenylalanines
A. Bertolani, L. Pirrie, L. Gazzera, A. Pizzi, G. Morra, M. Meli, G. Colombo, A. Genoni, G. Cavallo, G. Terraneo, P. Metrangolo
Chemistry a European Journal, (2017) 23: 2051-2058.
Thèmes scientifiques :
Nouvelles méthodes statistiques et quantiques pour la modélisation de systèmes biologiques complexes
Depuis presque quarante ans, la dynamique moléculaire, sorte de microscope informatique, est largement utilisée dans l’étude d’objets biologiques, formant ainsi un complément précieux des approches classiques de biologie structurale. De nombreux processus cellulaires restent cependant hors d’atteinte pour cette approche numérique, compte tenu des échelles de temps qu’ils couvrent. Au regard de la complexité des objets en jeu, différentes alternatives existent pour échantillonner les degrés de liberté caractéristiques les plus lents.
Nous développons des méthodes à la croisée des schémas d’échantillonnage d’importance et d’ensembles généralisés susceptibles d’offrir une solution convaincante aux limitations de la dynamique moléculaire. Dans le cadre du Laboratoire International Associé CNRS-UIUC , qui lie le CNRS à l’Université d’Illinois, ces développements sont portés dans les codes populaires NAMD et VMD, et sont utilisés dans l’étude de la thermodynamique de processus biologiques allant du changement conformationnel impliqué dans le transport membranaire aux phénomènes de reconnaissance et d’association de protéines


Le développement de nouvelles méthodes « à croissance linéaire » pour le raffinement de structures cristallographiques et de distributions électroniques dans des systèmes de grande taille (par exemple, protéines) est basé sur l’idée d’exploiter les bibliothèques d’orbitales moléculaires extrêmement localisées (ELMOs) que nous avons récemment construites.
Le but est de déterminer initialement des distributions électroniques approximatives pour les systèmes en examen et, après, en gardant fixes les orbitales transférées, d’optimiser les positions atomiques et les paramètres thermiques contre les facteurs de structure expérimentaux.
Croissance, communication cellulaire et réparation génomique

Nous nous intéressons au fonctionnement de processus biologiques fondamentaux à différentes échelles (du moléculaire au tissu) par des approches macro-, méso- et microscopiques. Nous étudions, en particulier, des problèmes concernant la mécanique des membranes biologiques ainsi que la croissance des tissus mous, comme les plantes ou encore les tissus cancéreux essentiellement à l’aide de la mécanique des interfaces fluides et élastiques. Jusqu’à présent les systèmes étudiés étaient en équilibre.
Nous modélisons également les filaments du cytosquelette des cellules et leurs interactions avec les membranes biologiques. Le comportement de ces filaments ne peut pas s’expliquer par la physique des polymères standard. Nous avons montré que des fluctuations conformationnelles internes doivent être prises en compte pour comprendre les microtubules. Cela a donné lieu à la notion de confotronique des biofilaments.
L’investigation par modélisation moléculaire de phénomènes qui contrôlent et modulent des processus clés des membranes biologiques impliqués dans la communication cellulaire est divisée en plusieurs thèmes. Ces travaux concernent le Laboratoire International Associé Pulsed Electric Fields Applications in Biology and Medicine. L’étude des phénomènes d’oxydation des membranes. L’oxydation des lipides des membranes biologiques est un processus clé qui influe sur les organismes vivants et le stress oxydatif des membranes.
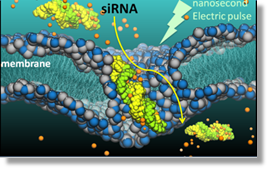

Elle est reliée à l’apparition de diverses maladies allant de l’inflammation de cancer et le vieillissement. L’ultime but de cette caractérisation est de nous permettre de proposer des nouvelles molécules anti-oxydantes membranaires efficaces. La vectorisation par ultrasons. Il s’agit d’utiliser des agents de contraste (microbulles) chargées ou non et des ultrasons pour perméabiliser les cellules avoisinantes. La caractérisation à l’échelle moléculaire de ce phénomène permettra le développement d’une technologie efficace pour la livraison ciblée de molécules.
Les canaux ioniques. L’objectif consiste à mettre en lumière les cascades physiopathologiques conduisant à des arythmies cardiaques et développer de nouveaux peptides pour moduler les canaux ioniques impliqués dans de telles cascades. Les transporteurs membranaires. A l’instar des canaux ioniques, ces protéines permettent d’importer ou d’exporter des espèces chimiques au travers de la membrane. Nous souhaitons ici décrypter la thermodynamique des changements conformationnels associés au fonctionnement de ces protéines pour mieux comprendre leurs dysfonctionnements et les pathologies qui leurs sont associées.


L’ADN est une cible biologique fondamentale au centre notamment des nouvelles techniques d’édition génomique ou encore des traitements par radiothérapie. Pour maintenir l’intégrité de l’information génomique, les cellules ont mis en place des stratégies pour réparer les lésions de l’ADN.
Pour mieux comprendre les mécanismes liés à l’endommagement et à la réparation, nous modélisons l’interaction de brins d’ADN, lésés ou non, avec des protéines de reconnaissance et de réparation. En particulier nous nous intéressons à la compréhension des facteurs structuraux et biophysiques (déformation de l’ADN, stabilité des complexes avec protéines) ainsi qu’à la réactivité sous-jacente, étudiée à l’aide de méthode QM et QM/MM, et à l’étude de la régulation épigénétique.
Collaborations principales :
Nationales
Université de Lorraine (CRM2, CRAN, L2CM, LORIA, IMoPA), ICS (Strasbourg), IBS, SyMMES (Grenoble), ENS (Lyon), IGR (Villejuif)
Internationales
MIT (Boston)
MRC MBU (Cambridge)
Cornell University (New York)
UIUC (Urbana-Champaign)
University of Chicago
Nankai University (Tianjin)
University of Bremen
Hong-Kong Chinese University
University of Pennsylvania
University of Washington
KAIST (South Korea)
